Zadanie 4.
Zadanie 4:
Implementacja procedur sekwencjonowania nowej generacji (NGS- next generation sequencing) celem pozyskania informacji do analiz filogenetycznych, które będą pozwalały na śledzenie dynamiki rozprzestrzeniania się wirusa i będą bezpośrednią podstawą do informowania o ogniskach zakażeń (clusters of transmission): szybka identyfikacja ognisk zakażenia, powiazań między nimi i szybka odpowiedź prewencyjna.
W ramach zadania opracowano i wdrożono technologię analiz filogenetycznych dla śledzenia dynamiki rozprzestrzeniania się SARS CoV-2. Na tej podstawie dokonano analizy filodynamicznej linii Sars-CoV-2 krążących pomiędzy przygranicznymi regionami polsko-niemieckimi z ujawnieniem sposobu rozprzestrzeniania się wirusa pomiędzy dwoma sąsiadującymi krajami europejskimi - Polską i Niemcami.
Dane molekularne pozwalają na implementację procedur sekwencjonowania nowej generacji (NGS- next generation sequencing) celem pozyskania informacji do analiz filogenetycznych, śledzenia dynamiki rozprzestrzeniania się wirusa. Należy podkreślić, że wnioskowanie filogenetyczne jest się popularną metodą śledzenia i monitorowania przenoszenia chorób wirusowych (HCV, HIV-1, Sars-CoV-2). Procedura opisuje precyzyjnie sposób porównania sekwencji do analiz filogenetycznych. W ramach prac na podstawie tej procedury dokonano analizy filodynamicznej linii Sars-CoV-2 krążących pomiędzy przygranicznymi regionami polsko-niemieckimi. Analiza ta opisuje rozprzestrzenianie się wirusa pomiędzy dwoma sąsiadującymi krajami europejskimi - Polską i Niemcami. Opracowanie obejmowało sekwencje wirusa notowane w 7 regionach: Pomorze Zachodnie, Lubuskie i Dolny Śląsk w Polsce oraz Meklemburgię-Pomorze Przedniego, Berlin, Brandenburgię i Saksonię w Niemczech wykazały odmienny regionalny wzorzec krążących linii Sars-CoV-2. Z wykorzystaniem epidemiologii molekularnej zbadano krążące pomiędzy dwoma sąsiadującymi krajami linie wirusa, aby zrekonstruować sieci transmisji Sars-CoV-2 (>13 000 sekwencji SARS CoV-2).
Począwszy od sierpnia 2020 r zaobserwowano wzrost dynamiki migracji wirusa na pograniczu polsko-niemieckim. Do końca maja 2021 na badanym regionie dominował jeden z czterech różnych kładów wirusa (20I(Alpha), 20E.EU1, 20A i 20B). Analiza filogeograficzna trzech fali epidemii umożliwiła zidentyfikować sieci transmisji najbardziej rozpowszechnionych wariantów wirusa krążących na pograniczu polsko-niemieckim. Zaobserwowano pięć dużych dwunarodowych klastrów dla linii innych niż alfa i dziewięć dla linii alfa. Niniejsze badanie prezentuje wzorce grupowania się różnych wariantów wirusa między zachodnią Polską a wschodnimi Niemcami (Rycina 3, 4). Śledzenie rozprzestrzeniania się wirusa między dwoma sąsiadującymi krajami umożliwia kierowanie polityką interwencyjną w transgranicznej współpracy dwustronnej przeciwko rozprzestrzenianiu się epidemii.
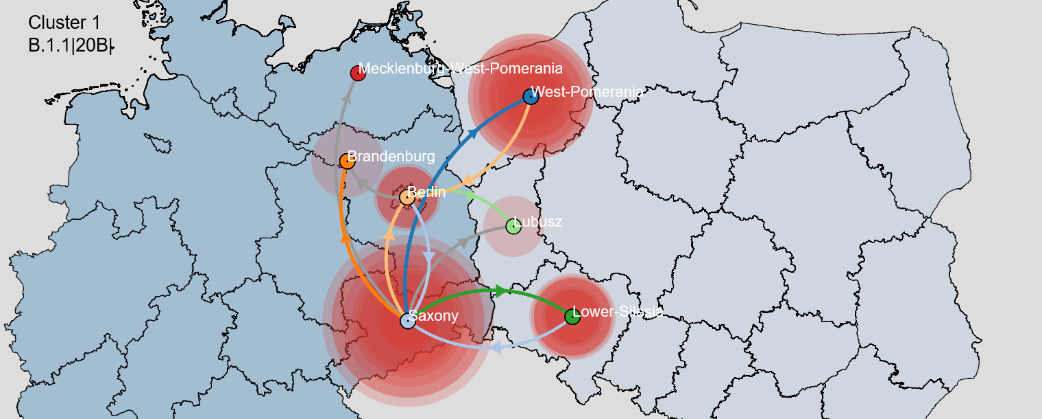
Rycina: Rekonstrukcja sieci transmisji największej zidentyfikowanej grupy wirusa wariantu innej niż Alfa (B.1.1) krążącej na pograniczu polsko-niemieckim.
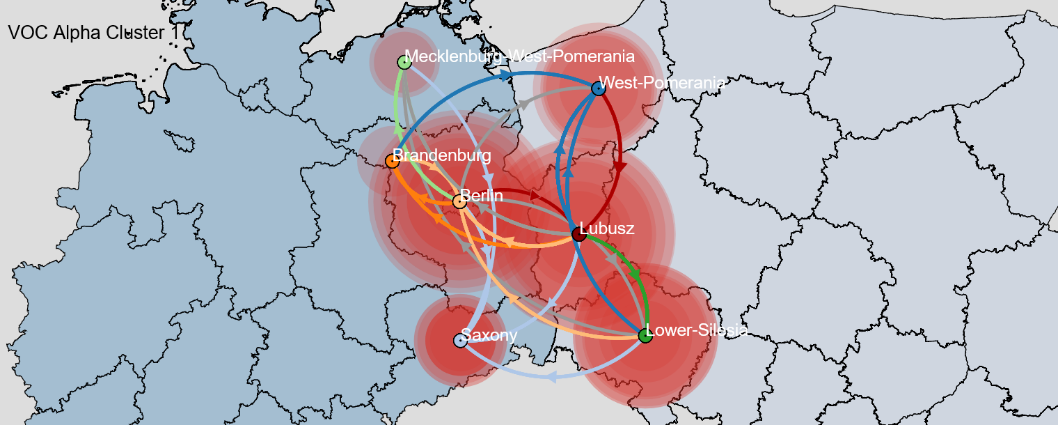
Rycina: Rekonstrukcja sieci transmisji największej zidentyfikowanej grupy wirusa wariantu Alfa krążącej na pograniczu polsko-niemieckim.
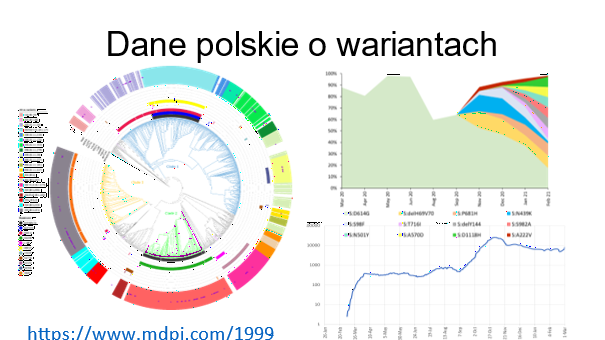
Rycina: Dane o wariantach krążących w pierwszych miesiącach pandemii
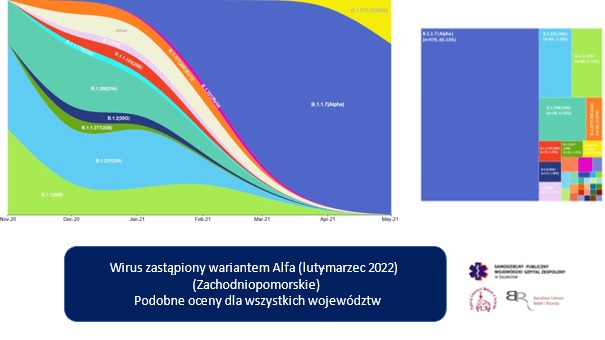
Rycina: Szybkość wymiany wariantów SARS CoV-2
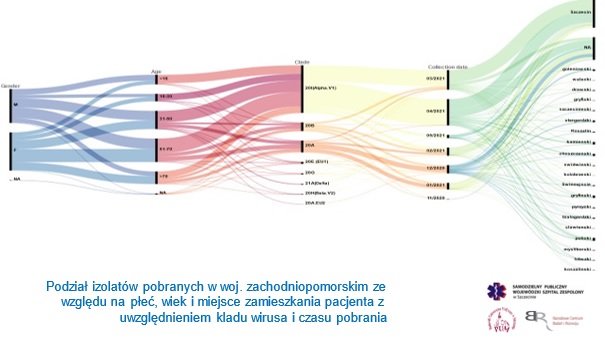
Rycina: Charakterystyka przepływu wariantów między grupami
Publikacje związane z zadaniem są dostępna pod adresem:
1. Serwin, K.; Aksak-Wąs, B.; Parczewski, M. Phylodynamic Dispersal of SARS-CoV-2 Lineages Circulating across Polish–German Border Provinces. Viruses 2022, 14, 884. https://doi.org/10.3390/v14050884
https://www.mdpi.com/1999-4915/14/5/884
2. Serwin K, Ossowski A, Szargut M, Cytacka S, Urbańska A, Majchrzak A, Niedźwiedź A, Czerska E, Pawińska-Matecka A, Gołąb J, Parczewski M. Molecular Evolution and Epidemiological Characteristics of SARS COV-2 in (Northwestern) Poland. Viruses. 2021 Jul 2;13(7):1295. doi: 10.3390/v13071295. PMID: 34372500; PMCID: PMC8310356.




